Julia spielt heute kaum eine Rolle, dabei überholt sie nicht nur im Wortsinn Python als Programmiersprache in Wissenschaft und Datenanalyse.

Python ist eine sehr beliebte Programmiersprache für Projekte im Bereich des Maschinellen Lernens und für viele andere wissenschaftliche Anwendungsgebiete - vor allem, weil man damit so einfach und schnell Prototypen programmieren kann. Doch die Ausführung von Python-Programmen ist sehr langsam. Die Programmiersprache Julia soll Python Konkurrenz machen, indem sie nicht nur einfach, sondern auch sehr schnell ist - fast so schnell wie C++.
Julia wurde 2012 vorgestellt und ist damit eine sehr junge Programmiersprache. Die Version 1.0 erschien im Jahr 2018.
Noch spielt Julia kaum eine Rolle: Bei den beliebtesten Programmiersprachen steht sie laut TIOBE-Index August 2021 auf Platz 26. Python hingegen liegt auf Platz 2 - mit guten Chancen, auch C noch zu überholen.
Nur eine einstellige Prozentzahl von Data Scientists und Machine Learning Engineers verwenden laut den Kaggle-Umfragen von 2017 bis 2020 Julia, Python hingegen über 80 Prozent. Doch Julia hat aufgrund der hohen Ausführungsgeschwindigkeit von Programmen gute Chancen, das zu ändern.
Julia: fast so schnell wie C
Python ist eine interpretierte Programmiersprache, die Python-Programme werden vor ihrer Ausführung also nicht kompiliert (in maschinennahen Code übersetzt), sondern während der Laufzeit interpretiert und ausgeführt. Deshalb sind interpretierte Programmiersprachen bei der Ausführung von Programmen oft langsam.
Das bedeutet nicht, dass alle Python-Programme langsam sind. Bei Python werden oft Bibliotheken, Frameworks beziehungsweise Pakete verwendet, um bestimmte Aufgaben zu erledigen. Bei Machine-Learning-Projekten wird eine Vielzahl solcher Pakete verwendet. Diese können, wenn die Ausführungsgeschwindigkeit wichtig ist, in anderem Programmiersprachen wie C++ programmiert sein.
Wenn man solche in C++ programmierte Komponenten verwendet, können auch Python-Programme schnell sein. Werden rechenintensive Algorithmen mit Python selbst programmiert, dann sind diese jedoch oft sehr langsam.
Zum Beispiel ist dies oft ein Problem bei Wissenschaftlern, die eine rechenintensive Simulation mit vielen Schleifen im Programm entwickeln. Solche Simulationen wären in Python in vielen Fällen sehr langsam. Doch viele Wissenschaftler, die keine Informatiker oder Vollzeitprogrammierer, sondern Experten ihrer jeweiligen Fachgebiete sind, meiden komplizierte Programmiersprachen wie C++.
Julia bietet sich hier als Alternative an. Sie ist vergleichbar benutzerfreundlich wie Python, jedoch eine Programmiersprache mit einem Compiler. Der Compiler übersetzt ein Julia-Programm in nativen Code der jeweiligen Plattformen (Betriebssysteme und CPUs) via LLVM, einer modularen Compiler-Infrastruktur.
Dabei handelt es sich um einen Just-Ahead-of-Time-Compiler (JAOT-Compiler). Das "Just" ist hier von großer Bedeutung. Im Gegensatz zu einem klassischen Ahead-Of-Time-Compiler (AOT-Compiler) wie in der Regel bei C++, wird bei Julia mit dem JOAT das kompilierte Programm nicht auf Dauer persistiert. Es existiert nur, solange der Julia-Prozess existiert. Wenn man ihn schließt, geht das kompilierte Programm verloren und muss beim nächsten Mal neu kompiliert werden.
Dies erfordert Zeit und beansprucht intensiv Hardwareressourcen wie CPU und RAM, weshalb bei Ad-hoc-Programmierung und Ausführung von sich in Entwicklung befindenden Programmen Julia eine höhere Anlaufzeit als Python hat. Nachdem ein Julia-Programm kompiliert wurde, ist es in vielen Fällen jedoch deutlich (manchmal um mehr als das 10- oder sogar 100-Fache) schneller als reine Python-Programme - solange man den Julia-Prozess nicht beendet.
An dieser Stelle sei noch darauf hingewiesen, dass es mit dem Packagecompiler.jl möglich ist, kompilierte Julia-Programme, die man gegebenenfalls etwas anpassen muss, dauerhaft zu persistieren und somit die Startzeiten zu verkürzen.
Nun zu den Benchmarks.
Die Gruppe Benchmarksgame präsentiert Ergebnisse mehrerer Benchmarks für Julia und Python auf ihrer Webseite: Bei einem n-body-Benchmark und auch beim Mandelbaum-Benchmark war Julia um mehr als das Hundertfache schneller als Python. Zum Vergleich: C (GCC) war weniger als doppelt so schnell wie Julia. Bei binären Bäumen erreichte Julia immerhin mehr als die sechsfache Leistung von Python. Dagegen war Julia beim Regex-Redux-Benchmark um etwa einen Viertel langsamer als Python.
Ergebnisse weiterer Benchmarks haben die Nasa-Mitarbeiter Jules Kouatchou und Alexander Medema in ihrem Artikel Basic Comparison of Python, Julia, Matlab, IDL and Java (2019 Edition) veröffentlicht. Die Zusammenfassung ist im Balkendiagramm am Ende des Artikels zu finden, wo ersichtlich ist, dass Julia ähnlich schnell wie C beziehungsweise Fortran und um ein Vielfaches schneller als Python ist.
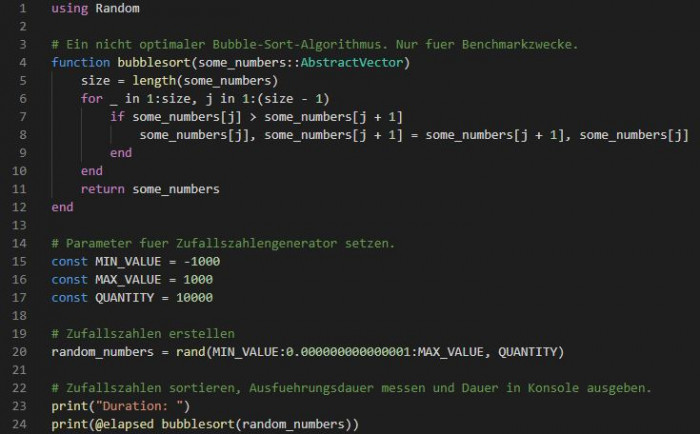
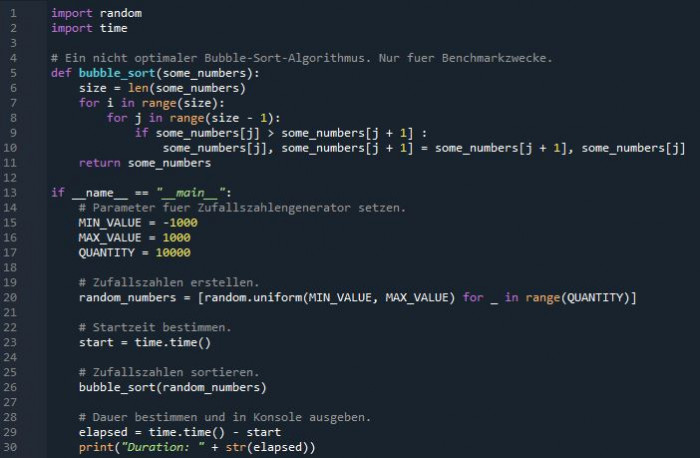
Einen kleinen Single-Core-Benchmark habe ich basierend auf dem Bubble-Sort-Algorithmus jeweils in Python und Julia programmiert. Hierbei habe ich auf spezielle, externe, in der Programmiersprache C entwickelte Bibliotheken verzichtet, um die Performance von Julia beziehungsweise Python selbst zu messen.
Außerdem sind die Bubble-Sort-Algorithmen nicht optimal programmiert. Auf eine Optimierung habe ich verzichtet, weil der Algorithmus nur für den Benchmark gedacht ist. Gemessen wird die Dauer für die Sortierung von 10.000 Zufallszahlen. Als System wurde ein PC mit einem AMD-Ryzen-5-2600-Prozessor, 16 GByte DDR4-3200-RAM und dem Betriebssystem Microsoft Windows 10 Pro Version 20H2 64 Bit mit Julia Version 1.6.2 und Python Version 3.8.11 verwendet. Bei diesem Benchmark ist Julia mit etwa 0,17 Sekunden mehr als 100-mal schneller als Python mit rund 22 Sekunden.
Die Benchmark-Programme können als Dateien heruntergeladen und die Ausführungsgeschwindigkeiten auf dem eigenen Computer ermittelt werden.
Die Ausführungsgeschwindigkeit von Programmen ist ein wichtiger Aspekt von Programmiersprachen, doch um eine hohe Beliebtheit für vielfältige Einsatzzwecke und Projekte zu erreichen, braucht eine Programmiersprache auch eine große Auswahl von hochwertigen Paketen beziehungsweise Bibliotheken. Schließlich will man nicht jedes Mal das Rad neu erfinden. Wie sieht hier die Situation bei Julia und Python im Vergleich aus?
Python ist rund drei Jahrzehnte alt und es existiert eine riesige Anzahl von Paketen in der Python-Repository. Aktuell (Stand September 2021) gibt es über 320.000 Projekte, davon mehr als 4.000 beziehungsweise mehr als 3.000 Projekte in den für Data Science besonders wichtigen Gebieten künstliche Intelligenz und Datenbanken. Als beispielhaften Stellvertreter für wissenschaftliche Gebiete habe ich die Astronomie und die Mathematik als Themen bei der Paketsuche gewählt und bekam über 1.000 Pakete für astronomische beziehungsweise über 2.600 Pakete für mathematische Themen gelistet.
Die Zahlen selbst sollte man nicht überbewerten, weil auch die Qualität der Pakete berücksichtigt werden muss. Obwohl es mehr als 4.000 Pakete in der Kategorie künstliche Intelligenz für Python gibt, verwenden Data Scientists vermutlich nur wenige Dutzend Pakete im Alltag. Viele Pakete sind nur Forks anderer Projekte oder begonnene, aber nicht mehr weiterentwickelte Programme oder sehr spezielle Nischen-Projekte.
Bezüglich der Anzahl von Paketen kann natürlich eine so junge Programmiersprache wie Julia mit ihren aktuell etwas über 6.000 Paketen nicht mit Python mithalten. Davon sind etwa 120 bis 130 Pakete dem Bereich künstliche Intelligenz zugeordnet. Etwa 30 Pakete gehören zur Kategorie Datenbanken. Es gibt 24 Pakete für das Fachgebiet Astronomie und einige Hundert Pakete im Bereich Mathematik. Eine komfortable Suche nach Julia-Paketen bietet die Webseite Juliahub.
Die Julia-Pakete in diesem Artikel vorzustellen, wäre ausufernd. Dennoch, um Data Scientists, die aus der Python-Welt kommen, den Einstieg in Julia etwas zu erleichtern: Ja, auch in Julia gibt es Dataframes und schöne Diagramme kann man schnell und einfach mit Plots erstellen. Plots stellt eine High-Level API bereit, die im Backend Frameworks wie zum Beispiel GR, PyPlot, PGFPlotsX und PlotlyJS verwendet. Einen visuellen Einstieg bietet die Julia Plots Gallery.
Unabhängig davon, wie benutzerfreundlich eine Programmiersprache und die APIs der verwendeten Frameworks und Pakete sind, die meisten Programmierer werden ungern mit simplen Texteditoren arbeiten. Gute Entwicklungsumgebungen (englisch: IDEs) sind für den Erfolg einer Programmiersprache von großer Bedeutung.
Entwicklungsumgebungen für Python
Bei Python gibt es nicht die eine dominierende IDE. Je nach Vorliebe und Einsatzweck haben sich mehrere IDEs etabliert.
Von Microsoft gibt es Visual Studio IDE und Visual Studio Code, die aber unter Data Scientists eher seltener Einsatz finden. Visual Studio IDE bietet sich eher für die Entwicklung von Anwendungen und Visual Studio Code für das Skripten mit Python an.
Eine Alternative für die Anwendungsentwicklung mit Python ist PyCharm von Jetbrains, welche für Linux, MacOS und Windows verfügbar ist. Auch diese IDE ist bei Data Scientist eher selten in Einsatz.
Data Scientist verwenden viel lieber die IDE Jupyter Notebook. Sie läuft im Browser und ist eine Art interaktives Notiz- oder Forschungsheft. Darin kann man sowohl Beschreibungen und Kommentare mit einer sehr einfachen Formatierungssprache verfassen als auch Python-Quellcode schreiben, ausführen und die Ergebnisse im Heft persistent halten.
Der große Nutzen für Data Scientists ist, dass sie sowohl den Weg, die Ergebnisse zu bekommen - nämlich die Formeln und den Quellcode - als auch die Ergebnisse selbst in Form von Beschreibungen, Ergebnistabellen, statistischen Werten und Diagrammen in einem sequenziellen Heft aufbewahren und ihren Kunden präsentieren können.
Zudem sind die Ergebnisse so replizierbar und bei Bedarf kann man einige Parameter im Python-Quellcode verändern und ausprobieren, ob man dadurch neue Erkenntnisse über die Daten bekommt beziehungsweise das Machine-Learning-Modell besser arbeitet.
Beispiele von Jupyter Notebooks bei Machine-Learning-Projekten werden auf Github gezeigt.
Will man jedoch nicht statistische Ergebnisse und Diagramme als Forschungsbericht präsentieren, sondern eine Machine-Learning-Anwendung entwickeln, dann ist oft Spyder (Scientific Python Development Environment) eine interessante IDE. Spyder zählt zu den beliebtesten Python-Entwicklungsumgebungen für Wissenschaftler. Sie ist relativ einfach, nicht überladen und verfügt über die wichtigsten Features wie einen komfortablen Debugger und Variablenmonitor.
Muss man sich unbedingt für eine bestimmte IDE entscheiden? Wenn man mehrere IDEs verwenden will, muss man jede einzeln installieren und sich um Updates kümmern? Muss man unterschiedliche Python-Versionen für unterschiedliche Projekte installieren und konfigurieren? Die Lösung all dieser Probleme ist der Anaconda Navigator.
Anaconda Navigator bietet die Installation von Jupyter Notebook, Spyder, Visual Studio Code und einigen anderen IDEs an - alles unter einer Haube samt grafischer Benutzeroberfläche. Zudem enthält der Anaconda Navigator einen benutzerfreundlichen Paketmanager für Python, mit dem man Pakete suchen, installieren und updaten kann. Auch das Updaten der IDEs selbst wird angeboten. Zudem bietet der Anaconda Navigator die Konfiguration mehrerer Environments an. Das sind unterschiedliche Umgebungen, unter denen der Python-Code laufen soll.
Man stelle sich zum Beispiel vor, dass man eine bestimmte Umgebung mit Tensorflow (ein Framework für die Entwicklung neuronaler Netze) und einer bestimmten Python-Version benötigt, weil vielleicht die aktuelle Version von Tensorflow nicht mit jeder Python-Version läuft; dagegen braucht man vielleicht für Keras (ein High-Level-Framework, das meistens Tensorflow im Backend verwendet) eine andere Python-Version oder man möchte unterschiedliche Cuda-Versionen ausprobieren. Statt Anaconda Navigator mehrmals zu installieren, installiert man ihn nur einmal und erstellt unter ihm mehrere Environments.
Wie man sieht, ist die Auswahl an IDEs bei Python groß. Doch wie sieht es bei Julia aus?
Obwohl Julia noch sehr jung ist, gibt es schon mehrere Entwicklungsumgebungen zur Auswahl. Die folgende Übersicht berücksichtigt nicht alle existierenden IDEs, sondern nur jene, die aus meiner Sicht und Erfahrung bei wissenschaftlichen Projekten, zu denen auch Data-Science-Projekte zählen, eine große Rolle spielen.
Noch bis vor kurzem war Juno, eine Erweiterung für den Editor ATOM, die wohl beliebteste Entwicklungsumgebung für Julia. Juno hat sehr viele Funktionalitäten und ist sehr mächtig. Doch, wie in einem Video der Juliacon 2020 mitgeteilt wurde, hat sich das Juno-Entwicklerteam entschieden, die Weiterentwicklung von Juno für ATOM zu stoppen und sich auf die Entwicklung der Julia-Erweiterung für Visual Studio Code zu konzentrieren.
Im Video wird als ein Grund dafür die Verlangsamung der Weiterentwicklung des Editors ATOM genannt. Vielleicht gibt es auch andere Gründe. Meiner Erfahrung nach war Juno für ATOM zwar sehr mächtig, aber bei Updates gab es oft Kompatibilitätsprobleme zwischen verschiedenen Paket-Versionen, die mich mehrmals dazu zwangen, die Software zu deinstallieren und neu zu installieren. Dies führte dazu, dass ich Angst hatte, ATOM oder Juno auf eine neue Version zu aktualisieren und bewusst länger ältere, stabile Versionen verwendet habe.
Nichtsdestotrotz kann man Juno mit ATOM weiterhin nutzen, aber es wird keine Weiterentwicklung mehr geben. Die Zukunft gehört der Julia-Erweiterung für Visual Studio Code.
Visual Studio Code ist von Microsoft und für die Betriebssysteme Windows, Linux und MacOS verfügbar. Es gibt eine riesige Anzahl von Erweiterungen für Visual Studio Code, um es als Entwicklungsumgebung für eine Vielzahl von Programmiersprachen zu verwenden. Informationen und eine Dokumentation über die Julia-Erweiterung für Visual Studio Code findet man auf der Webseite Julia-vscode.org.
An dieser Stelle möchte ich insbesondere auf die Dokumentation zum Starten eines Julia-Programms in Visual Studio Code hinweisen. Manche Julia-spezifischen Funktionalitäten wie die Darstellung von Diagrammen werden nur im REPL-Modus (Real-Eval-Print-Loop Command Line von Julia) unterstützt. Um Julia-Programmcode in Visual Studio Code im REPL-Modus zu starten, muss man die Tasten Steuerung (Ctrl) und Eingabe (Enter) gleichzeitig drücken. Zum Ausführen von Code-Blöcken oder Code-Zellen gibt es die Tastenkombinationen Alt und Enter oder Shift und Enter. Mehr Informationen findet man in einer Dokumentation.
Juno für ATOM und die Julia-Erweiterung für Visual Studio Code bilden Entwicklungsumgebungen, die für die allgemeine Entwicklung von Programmen mit Julia gut geeignet sind. Doch Data Scientists präferieren oft sogenannte Notebooks wie zum Beispiel die IDE Jupyter Notebook, die kurz vorgestellt wurde. Fans von Jupyter Notebook können sich freuen: Man kann statt Python auch Julia-Programme in Jupyter Notebook ausführen. Wie man das macht, erfährt man beim Projekt JuliaLang/IJulia.jl in Github und bei einer Schritt-für-Schritt Anleitung bei Data to Fish.
Jupyter Notebook ist keine native Entwicklungsumgebung für Julia, sondern ist für unterschiedliche Programmiersprachen gedacht und wird hauptsächlich von Python-Entwicklern verwendet. Doch es gibt auch Notebook-Entwicklungsumgebungen speziell für Julia.
Pluto ist eine Notebook-Entwicklungsumgebung, die speziell für Julia und mit Julia entwickelt wurde und wie Jupyter Notebook im Browser läuft. Pluto wurde hauptsächlich mit Julia programmiert. Das Frontend von Pluto benutzt Javascript und CSS.
Wichtige Besonderheiten von Pluto sind:
1. Pluto ist reaktiv, das heißt, sobald man in einer Code-Zelle den Wert einer Variablen verändert, prüft Pluto alle Abhängigkeiten im Programmcode und führt alle abhängigen Code-Zellen automatisch aus.
2. Pluto ignoriert die Reihenfolge der Code-Zellen. Man kann eine Funktion definieren, die zum Beispiel eine Berechnung abhängig von der Variablen x durchführt und ein Diagramm erstellt. Der Variable x kann man auch danach, das heißt in einer darauffolgenden Code-Zelle, einen Wert geben. Pluto erkennt die Abhängigkeiten und führt im Hintergrund das Programm in sinnvoller Reihenfolge aus. Das bedeutet aber auch, dass man standardmäßig nur an einer Stelle im Programm einer globalen Variablen einen Wert zuweisen darf, sonst kommt es zu einer Fehlermeldung.
Die Idee, so zu programmieren, kommt aus dem Alltag von Mathematikern und Naturwissenschaftlern und vermutlich erinnern sich manche von uns an ihre Schul- und Studienzeit: Oft definiert man erst eine Funktion wie y = x² - 2x + 4, um eine Aufgabe zu lösen und erst danach setzt man Werte für die Variablen ein und berechnet das Ergebnis.
3. Pluto-Notebooks werden als gewöhnliche Julia-Dateien gespeichert und können in anderen IDEs wie zum Beispiel Visual Studio Code geöffnet und ausgeführt werden. Im Gegensatz zu Jupyter Notebook gibt es bei Pluto nur Code-Zellen und keine Dokumentationszellen (Markdown Cells). Dokumentationen im Wiki-Stil schreibt man bei Pluto indem man in der Julia-Code-Zelle die Markdown-Bibliothek mittels "md" für Julia verwendet.
4. Standardgemäß akzeptiert eine Code-Zelle nur einen Befehl. Wenn man mehrere Befehle in einer Code-Zelle verwenden möchte, muss man in der Code-Zelle einen Block beginnend mit "begin" und endend mit "end" definieren. Im Block darf man dann mehrere Befehle verwenden.
5. Während man programmiert und Befehle eingibt, bekommt man in Echtzeit eine Dokumentation zu den Befehlen, die man verwendet (Live docs).
Es gibt Data Scientists, die Pluto toll finden, aber dessen reaktives Verhalten nicht mögen, weil dies unter Umständen viel Rechenleistung erfordert und man in manchen Fällen den Wert einer Variablen verändern will, ohne alle abhängigen Programmteile sofort ausführen zu wollen. Hier bietet sich Neptune als Alternative an. Neptune ist ein Fork von Pluto, der viele Funktionalitäten von Pluto hat, aber nicht reaktiv ist und bei dem globale Variablen in mehr als einer Code-Zelle Werte zugewiesen bekommen dürfen. Ein weiterer Unterschied zu Pluto ist, dass Neptune standardmäßig mehrere Befehle in einer Code-Zelle akzeptiert. Ein Begin-end-Block ist nicht erforderlich.
| Merkmal | Jupyter Notebook | Neptune Notebook | Pluto Notebook |
|---|---|---|---|
| Dateiformat | JSON | Julia Quellcodedatei | Julia Quellcodedatei |
| Mehrere Befehle in einer Code-Zeile? | Ja | Ja | Mit begin-end-Block |
| Programmiert in | Hauptsächlich Python | Hauptsächlich Julia | Hauptsächlich Julia |
| Programmiert für | Hauptsächlich Python, R und Julia. Außerdem für viele weitere Programmiersprachen über Drittanbieter. | Julia | Julia |
| Reaktiv | Nein | Nein | Ja |
| Realtim-Documentation | Nein | Ja (Live docs) | Ja (Live docs) |
| Wertzuweisung an globale Variablen | Mehrmals erlaubt | Einmalig | Mehrmals erlaubt |
| Zellenarten | Code, Markdown, Raw | Code (Markdown via md-Command) | Code (Markdown via md-Command) |
| Link | https://ift.tt/3FLN6yW | https://ift.tt/3iXoEkc | https://ift.tt/2QVqHaw |
Stand 12. September 2021 ist die aktuelle, stabile Version 1.6.2. Julia steht kostenlos als Open-Source-Programmiersprache für FreeBSD, Linux, MacOS und Windows zum Download bereit.
Fazit
Obwohl Julia eine noch sehr junge Programmiersprache ist, existieren schon viele Entwicklungsumgebungen. Python-Programmierer müssen auf die gewohnten IDEs Visual Studio Code und Jupyter Notebook nicht verzichten - diese gibt es auch für Julia.
Alternativ gibt es noch die Notebook-IDEs Neptune und Pluto, die beide in und für Julia programmiert wurden und innovative Besonderheiten haben. Beide verwenden Julia-Quellcodedateien als Dateiformat und haben eine Echtzeitdokumentation als Unterstützung für den Programmierer. Zudem ist Pluto reaktiv, d.h. es reagiert auf Änderungen von Wertzuweisungen an Variablen in einer Code-Zelle, erkennt Abhängigkeiten und führt automatisch alle abhängigen Code-Zellen aus.
Wegen des Just-Ahead-of-Time-Compilers (JAOT) führt Julia in vielen Fällen Programme sehr viel schneller als Python aus. Benchmarks zeigen, dass Julia bezüglich der Ausführungsgeschwindigkeit in etwa auf dem Niveau von der Programmiersprache C++ liegt und somit in einer ganz anderen Performance-Klasse als Python spielt. Jedoch dauert das Starten eines Julia-Programms oft viel länger als eines Python-Programms, weil die verwendeten Komponenten vor der Ausführung kompiliert werden müssen.
Somit eignet sich Julia für Ad-hoc-Reporting und Ad-hoc-Datenanalyse, die Data Scientists insbesondere in den ersten Iterationen eines Projekts machen, nicht so gut wie Python. Julia ist dann vorteilhaft, wenn die Ausführungsgeschwindigkeit eine große Rolle spielt. Beispiele dafür sind Simulationen, die Berechnungen in vielen Schleifen im Programmcode enthalten, oder wenn der Programmierer mit Julia einen Deep- bzw. Machine-Learning-Algorithmus selbst entwickelt, statt externe Komponenten wie Tensorflow zu verwenden.
Auch wenn Julia bei der Zahl der Pakete noch lange nicht mit Python mithalten kann: Sie hat das Potenzial zu einer der wichtigsten Programmiersprachen in den Bereichen Naturwissenschaften und Data Science zu werden.
Miroslav Stimac ist Informatiker, promovierter Wirtschaftswissenschaftler und aktuell Teilzeitstudent der Astronomie. Er arbeitet seit 2004 als Entwickler mit branchenübergreifender Projekterfahrung, hauptsächlich in Business Intelligence, Data Science und Statistik. Seine fachlichen Interessenschwerpunkte sind Astronomie, Japanologie, Konsumforschung und Robotik.
Programmiersprache Julia: Wie Python, nur schneller - Golem.de
Read More

No comments:
Post a Comment